Un nouveau laboratoire pour une recherche inédite
Solange Cassette (SC) : 1981, la construction[1] du «Laboratoire de Biologie Moléculaire des Relations Plantes Micro-organismes[2]» est achevée, les personnels s’installent. Sa création découle du projet conjoint de Jean Dénarié et Pierre Boistard pour, je cite, « rassembler dans un même site les gens qui faisaient des études de génétique au niveau cellulaire […] tous au départ des microbiologistes. La thématique […] était les relations entre les plantes et les micro-organismes» [3].
La même année, vous obtenez votre BTS Analyses biologiques à l’ENFA[4] de Toulouse et deux ans plus tard, en mars-1983, vous êtes recrutée à l’INRA de Toulouse. Qu’est-ce qui vous a poussée à postuler pour venir travailler à l’INRA ?
Fabienne Maillet (FM) : Mon BTS avait une composante agronomique. J’ai fait mon stage à l’INRA, suivi d’un CDD de 3 mois dans le même service, mais il a fallu du temps avant que des postes ne s’ouvrent sur le centre. J’étais en CDD à l’Institut Pasteur de Nouméa où vivaient mes parents quand j’ai reçu une lettre indiquant que deux postes étaient créés dans cette nouvelle unité de l’INRA. Mon dossier avait été sélectionné : hypermotivée, je suis rentrée immédiatement pour passer l’entretien oral. J’ai été recrutée en tant que contractuelle puisque nous n’étions pas sous statut de fonctionnaire à l’époque.
SC : Vous intégrez, au grade 2B[5], l’équipe de Jean Dénarié qui travaille sur la fixation de l’azote par les plantes en symbiose avec des bactéries[6]. Pouvez-vous nous raconter votre arrivée dans cette équipe ? Saviez-vous sur quel thème vous alliez travailler, en quoi consiste votre travail à ce moment-là ?
FM : Je connaissais deux membres de l’équipe car à l’entretien il y avait Jean Dénarié et Charles Rosenberg qui m’ont un peu parlé de la thématique. Je suis arrivée « comme un papillon sur une fleur ». Je ne savais pas trop ce qui m’attendait, si j’allais faire uniquement de la microbiologie ou si j’allais découvrir autre chose. J’ai été très bien accueillie et encadrée par M. Rosenberg. Jean Dénarié était plutôt dans les sphères d’instruction du sujet et Charles était plus sur le versant expérimentation. L’équipe était très réduite à cette époque ; je ne sais même plus s’il y avait des étudiants quand je suis arrivée. Il y avait deux autres techniciennes arrivées un peu avant moi et de mon âge. Je me suis vite intégrée et régalée à « bidouiller » les microbes (rhizobium) mais aussi les plantes (luzerne) : notre sujet était l’interaction entre les deux. A cette époque, notre objectif était de trouver les gènes de la bactérie nécessaires à l’établissement de cette interaction. Nous devions cultiver les plantes puis les inoculer avec des bactéries sauvages ou mutées et voir leurs réactions. Ces deux domaines (génétique bactérienne et culture végétale in vitro) m’ont de suite été attribués.
L’interdisciplinarité avec les cytologistes et les biochimistes
SC : L’équipe de Jean Dénarié travaille en interdisciplinarité avec des cytologistes du LBM, dont Georges Truchet et des biochimistes du CRBGC[7], Jean-Claude Promé en particulier. Quelles sont les interactions avec eux dans votre travail à cette époque ?
FM : En évoquant cette découverte, je pense que cela n’aurait pas pu se faire sans la réunion de ces trois équipes. Georges Truchet était à Marseille quand on a commencé les travaux. Lui et Jacques Vasse ont tout fait pour venir sur Toulouse, intégrer l’équipe et ainsi être au plus près. Cela a boosté les choses c’est évident ! A la suite, c’était des interactions permanentes : en fin de journée, souvent un chercheur allait dans le bureau de l’autre et discutait longuement. Je le vois moins maintenant car nous sommes très nombreux et ce sont des réunions plus formelles. C’est vrai que c’était un petit monde à l’époque : nous étions peu, localisés dans un seul bâtiment, il était très facile de communiquer[8].
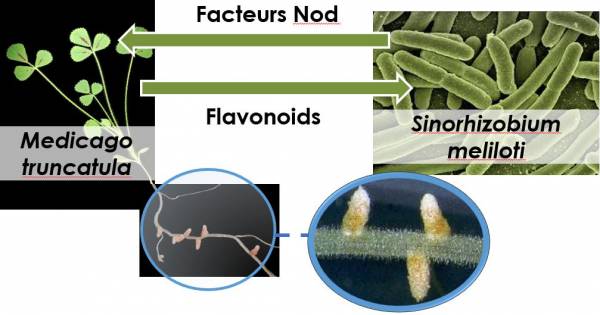
L’équipe de Georges Truchet a mis au point un test biologique qui permettait de suivre une activité spécifique des bactéries puis des surnageants bactériens sur la plante. L’équipe de Jean-Claude Promé, en biochimie, était chargée de purifier ce surnageant bactérien pour essayer d’isoler la molécule qui avait cette activité. Mon rôle et celui de Catherine Faucher alors doctorante, consistait à faire des tests biologiques pour l’équipe de cytologie, à préparer des surnageants bactériens pour que l’équipe de biochimistes puisse faire les différentes étapes d’extraction. Au fur et à mesure que les choses se sont précisées, j’ai été plus impliquée dans les méthodes d’extraction. Je fournissais des échantillons de plus en plus purs pour faire les tests biologiques pour lesquels Georges Truchet faisait la dernière lecture. Par ce moyen, on est arrivé à isoler une fraction active du surnageant assez pure. Ensuite les biochimistes ont travaillé d’arrache-pied pour trouver la composition et la structure exactes de ce petit mélange de molécules qu’on appellera désormais Facteurs Nod.
SC : Patrice Lerouge nous a raconté cette histoire de la découverte des facteurs Nod, il a fait appel à sa mémoire[9]. Vous aviez surement des contacts avec lui, qu’est-ce que vous pouvez nous en dire ?
FM : L’équipe de biochimistes était organisée un peu comme chez nous. Il y avait Jean-Claude Promé à la tête du groupe, Patrice Lerouge –alors jeune chercheur qui a eu la médaille du CNRS pour ces travaux- était plus proche de nous techniciens et étudiants, et enfin Philippe Roche qui préparait sa thèse. Chez nous, il y avait Catherine Faucher qui passait sa thèse au moment de la découverte de la molécule. C’était des interactions à tous les niveaux : on se rassemblait quelquefois tous ensemble et on travaillait entre « paillassistes ». On est allé les voir pour se former. Ils sont venus au laboratoire où ils nous ont conseillés notamment pour l’achat d’une chaîne HPLC, un gros appareil pour séparer les molécules à l’intérieur d’un mélange. On a eu effectivement beaucoup d’interactions et jusqu’au bout. Après, chacun a fait son chemin.
J’ajoute que j’ai toujours été en position charnière, c’est-à-dire à l’interface entre les trois équipes, mon travail de génétique bactérienne consistant à construire des souches bactériennes capables de fournir du surnageant très actif et mon apprentissage en biochimie pour l’extraction et la purification de la molécule. La réalisation des tests biologiques a permis de suivre l’activité, orienter les méthodes d’extraction et repérer l’activité dans les différentes fractions. En fait, j’ai toujours été au milieu de ce triangle-là.
Découverte des facteurs Nod et publication scientifique majeure
SC : Il y a 30 ans, en avril-1990, paraît dans la revue Nature la publication sur la découverte des facteurs Nod[10]. Vous êtes co-publiante. Avec des mots accessibles pour tous, de quoi s’agit-il ?
FM : En mots accessibles, c’est dire que les équipes de chercheurs réunis à ce moment-là ont découvert la structure de la molécule qui était la clé de la bactérie symbiotique pour se faire reconnaître et rentrer dans le système racinaire de sa plante-hôte afin d’établir la symbiose fixatrice d’azote.
La symbiose est une association entre deux organismes qui est bénéfique pour les deux à la fois. Dans notre cas, la bactérie s’associe avec la plante au niveau racinaire, dans un nouvel organe appelé nodule, pour transformer l’azote minéral de l’air en ammoniac directement utilisable par la plante. A l’inverse, la plante fournit les composés carbonés pour le métabolisme de la bactérie. C’est vraiment une interaction à bénéfices mutuels.
SC : Et l’aventure scientifique ? Est-ce que vous aviez conscience de cela ?
FM : Je savais qu’il y avait un gros challenge qui avait été réussi et cela m’avait enchantée. Ce que j’avais trouvé puissant est que c’était la réunion, le travail et l’interaction de ces trois équipes de disciplines très différentes qui avaient permis ce succès. Nous avions fait une grande avancée dans la compréhension de ce système symbiotique pour répondre à la question : « Comment une plante peut-elle accepter l’intrusion d’une bactérie ? » C’était grâce à ce signal, cette petite molécule qu’on avait trouvée et qui avait, en plus, une structure très atypique pour être synthétisée par une bactérie. C’était une molécule qu’on aurait très bien vue fabriquée par un champignon car elle a une structure chitinique[11]. Chose très inattendue de la part d’une bactérie de synthétiser un tel composé.
En fait, j’ai été frappée par deux aspects : d’abord l’avancée dans la compréhension de la symbiose pour laquelle j’avais été recrutée, et aussi que ce soit une découverte inattendue car cela avait vraiment une structure inédite. Cette molécule n’était pas répertoriée dans les catalogues chimiques de l’époque.
SC : Sur les clauses de confidentialité, étiez-vous tenue au secret ?
FM : A l’époque non, en tout cas, je n’en ai pas eu conscience. En revanche, lorsqu’on s’est aperçu que cette molécule était inédite, qu’elle avait des activités sur plante intéressantes, à partir du moment où Jean Dénarié a décidé de breveter cette molécule, oui il y a eu des consignes. Et c’est surtout après, lors du déroulement des contrats de recherche signés avec notre partenaire industriel, que j’ai senti les contraintes de confidentialité ; elles n’ont pas toujours été bien perçues. Des chercheurs étaient arrivés ; il y avait des sous-thématiques un peu différentes des nôtres, et nous dans le groupe « signaux » étions assez contraints, même par rapport à nos proches collègues.
SC : Justement, suivent deux brevets déposés en 1990 et 1992 où vous êtes mentionnée « Inventor ». Qu’est-ce que cela change professionnellement pour vous ? Que ressentez-vous à ce moment ?
FM : Cela ne change rien du tout professionnellement, disons pas plus que la publication. J’avais l’impression de faire partie d’une équipe qui avait fait des choses intéressantes et qui pouvaient déboucher sur une application. Lorsqu’on dépose un brevet, on espère que cela intéressera une entreprise qui l’utilisera pour une application commerciale, scientifique, agronomique, médicale… Il est vrai que cette perspective est gratifiante. C’est l’INRA[12] qui s’est occupé des démarches pour le brevet et cela a été relativement long.
SC : La suite en 1997-1998 est le début du séquençage des génomes des bactéries Rhizobium et Ralstonia[13]. Quelle est votre participation ?
FM : Aucune car l’équipe s’était déjà bien étoffée et il y avait des purs généticiens moléculaires qui s’en étaient occupé, notamment Frédéric Debellé.
Pour ma part, je travaillais sur le versant « applications des facteurs Nod ». Avant le dépôt de brevet, on a réalisé des quantités d’expériences avec des facteurs Nod purifiés pour voir quelles réactions pouvait provoquer cette molécule vraiment pure. On s’est aperçu qu’il y avait des retombées intéressantes : cela permettait un meilleur développement racinaire, une mise en place de la symbiose un peu plus rapide. C’est alors qu’on a pensé qu’il pouvait y avoir une application agronomique à l’utilisation de cette molécule, d’où le brevet.
Mise en application de la découverte : test en champs, mises au point, commercialisation
FM : Une fois le dépôt de brevet acté, il a fallu chercher et prendre des contacts avec des sociétés pouvant être intéressées. C’est, quasiment à 100%, Jean Dénarié qui s’en est occupé. Cela n’a guère intéressé de sociétés françaises. C’est une société américaine qui a achetée l’option de licence. Il a fallu alors faire un travail de mise au point du procédé de purification pour en produire beaucoup[14] et ainsi faire des tests en champs. Puis des mises au point pour validation des produits commerciaux et enfin former des ingénieurs de la société.
SC : Oui, en 2000 débute la collaboration du LIPM avec la Sté NITRAGIN sur les inoculants bactériens. «Des millions d’hectares ont été et sont actuellement inoculés chaque année dans le monde avec différentes espèces de Rhizobia, dont environ 50 millions pour le soja». [15]
Une vraie réussite scientifique et industrielle avec l’application directe sur les cultures. Qu’en pensez-vous ?
FM : A l’époque, j’étais extrêmement fière et contente qu’une petite molécule comme cela puisse aider, faire évoluer vers une agriculture durable en utilisant moins d’engrais. C’était vraiment le but. C’est aussi pour cela que j’étais contente d’avoir intégré cette équipe. Au fil des années, il y a eu des critiques sur les statistiques faites par l’entreprise sur ces essais en champ, jugés peu efficaces. D’autres qui disaient que cela enrichissait surtout la société commerciale plutôt que l’agriculteur. Selon moi, l’agriculteur trouvait un intérêt car il achetait une première fois, et rachetait l’année suivante cet inoculant. Au départ, j’étais très enthousiaste, ensuite plus modérée. D’autant que cela n’est pas très développé en Europe, en France en tout cas.
SC : Est-ce que vous trouvez cela regrettable ?
FM : Oui, parce que c’est une recherche française, qui est peu appliquée en France et, par certains côtés, cela me perturbe un peu. De fait, c’est surtout le soja qui en a bénéficié et on n’est pas très cultivateur de soja en France[16]. C’est surtout le pois, qui a naturellement son rhizobium dans le sol, contrairement au soja qui n’est pas du tout issu des terrains américains et n’avait pas son rhizobium dans le sol, alors il fallait le lui apporter. En ajoutant en plus la molécule facteur Nod l’effet était plus net, plus sensible.
SC : Sur quelles cultures pourrait-on l’appliquer en France ?
FM : Le soja bien sûr car depuis, il y a eu un effort pour le cultiver. Concernant le pois, comme il a été introduit et cultivé en France depuis très longtemps, il peut trouver dans le sol ce qu’il lui faut, même si on peut toujours améliorer les choses et des recherches sont faites dans ce sens. Ensuite, en cultures de légumineuses, il y a la lentille, l’arachide mais ce n’est pas cultivé ici, le pois chiche -je ne sais pas si c’est très cultivé ici-, le trèfle pour la production fourragère, le haricot…. Vraiment, il y a de quoi faire !
La relation chercheurs—ingénieurs : singularité et complémentarité
SC : Selon Pierre Boistard, « La différence entre chercheurs et ingénieurs, c’est un état d’esprit différent. Le chercheur pose des questions, l’ingénieur a plus une vue synthétique, plus globale pour mobiliser des connaissances ; ce qui demande tout autant de qualités intellectuelles mais ce ne sont pas les mêmes »[17] Vous qui êtes ingénieure, quelle est votre position ?
FM : Je suis assez d’accord avec Pierre Boistard. L’ingénieur a-t-il une vue plus globale ? Je ne sais pas. Moi je dirais plus pratique en tout cas pour mettre en application l’idée, voir les outils qui sont nécessaires, essayer de valider l’idée, ou dire « Non, c’est une idée qui était fausse ». L’ingénieur est celui qui connaît les outils ou cherche d’autres outils qui permettent de répondre à la question que pose le chercheur. Selon moi, c’est cela la définition de l’ingénieur : trouver l’outil pour répondre à la question du chercheur.
Un parcours professionnel remarquable
SC : Vous êtes passée de Technicienne à Ingénieure d’Etudes hors classe, une belle réussite professionnelle. Votre carrière professionnelle est indissociable de ce laboratoire INRA-CNRS. Qu’en retenez-vous principalement ?
FM : Je dirais même qu’elle est indissociable de mon équipe car je n’ai jamais changé de groupe contrairement à d’autres techniciens et ingénieurs qui eux ont changé pour voir autre chose, aller ailleurs. Je suis restée car cette équipe a réussi à évoluer et à ajouter des thématiques et des technologies au fur et à mesure que le temps passait et que les recherches avançaient.
Quitte à me répéter, j’ai commencé par la bactériologie, la génétique bactérienne, j’ai ajouté la biochimie des glycolipides, la génétique végétale et tout cela a fait que dans cette équipe, j’ai évolué, j’ai appris quantité de techniques différentes et j’en apprends encore. En ce moment je m’oriente vers les aspects plus moléculaires. Ma carrière a été très évolutive : j’ai progressé, appris des choses nouvelles, agrandi mon domaine de connaissance.
SC : Il y avait un intérêt constant dans votre travail et des nouveautés continuelles. Il faut avoir cette capacité à intégrer…
FM : C’est vrai, il a fallu que j’aille me former auprès de ceux qui savaient. J’ai toujours préféré cette démarche plutôt que des formations très théoriques que j’ai toujours du mal à mettre en application ensuite. Je préfère aller passer du temps avec les biochimistes, auprès de leurs matériels. Actuellement j’apprends auprès des spécialistes de l’unité ; je vais les voir, les solliciter.
Épilogue
FM : J’ai vécu cette expérience en découvrant que le fruit d’une recherche pouvait être le résultat d’interactions entre des équipes de disciplines très différentes. J’étais toute jeune et je voyais qu’on pouvait faire de grandes choses en unissant les forces scientifiques et en mettant aussi en avant le travail des étudiants pour leur permettre d’avoir un beau diplôme ! Pour tout cela, c’était gratifiant !
J’ajoute que cette découverte a permis à beaucoup de chercheurs, -non seulement en France mais aussi à l’international-, de développer des quantités de projets et cela aussi est gratifiant. Actuellement, ce sont de tous petits maillons de la chaîne génétique et moléculaire qu’il faut caractériser un par un. Bien entendu, il y a énormément de choses à faire encore ; seulement c’est par petites étapes, de longue haleine et moins impressionnant désormais, sauf si un jour on arrive à faire fixer l’azote de l’air par une céréale.
-
- [1] Décision prise en 1978 par Jacques Poly, Directeur Général de l’INRA et André Berkaloff, Directeur du Département Sciences de la Vie au CNRS de créer, sur le campus INRA d’Auzeville-Tolosane, cette UMR INRA-CNRS.
- [2] LBMRPM : devient ensuite le LIPM – Laboratoire des Interactions Plantes Micro-organismes
- [3] Source : Pierre BOISTARD, Archorales, Les métiers de la recherche : témoignages, INRA Tome 12, 28-avril-2005, p. 64
- [4] Ecole Nationale de Formation Agronomique de Toulouse
- [5] Actuel corps de Technicien
- [6] Source : Véronique SAINT GES. Facteurs Nod et Myc : des molécules signal aux champs pour un gain de productivité des cultures, Executive Summary. INRA, ASIRPA, mars-2014.
- [7] CRBGC : Centre de Recherche en Biochimie et Génétique Cellulaires. (devient ensuite le LPTF - Laboratoire de Pharmacologie et Toxicologie Fondamentale)
- [8] Source : Pierre BOISTARD, op cit. p. 68 « une organisation spatiale avec un centre de rencontre au milieu du laboratoire »
- [9]Voir témoignage de Patrice Lerouge
- [10] Source : Lerouge P, Roche P, Faucher C, Maillet F, Truchet G, Promé J-C, Dénarié J. (1990) Symbiotic host‐specificity of Rhizobium meliloti is determined by a sulphated and acylated glucosamine oligosaccharide signal. Nature, 344: 781‐784.
- [11] Note de F. Maillet : « Comme la paroi des champignons ou la carapace des crustacées »
- [12] INRA Transfert
- [13] Source : P. Boistard, op cit. p.65
- [14] Développement technique réalisé par la suite par le laboratoire CERMAV à Grenoble (UPR CNRS 5301)
- [15] Source : Cécile REVELLIN, UMR Agroécologie Dijon. Les symbioses fixatrices d’azote, Actes du colloque « Alliance au pays des racines », Paris - 25-mai-2012, p.32.
- [16] Cf. accord UE/USA sur le soja qui limite la production en Europe. Article du 27-08-2018 dans la revue La France Agricole
- [17] Source : Pierre Boistard, op cit. p. 74